Criando boxplots apresentáveis no R [Portuguese text]
Aprenda a gerar múltiplos boxplots e a organizá-los em uma única figura de alta qualidade.
Neste tutorial você aprenderá a gerar múltiplos boxplots e a organizá-los em uma única figura de alta qualidade.
Esta postagem foi inteiramente produzida em ambiente R Studio, usando os pacotes rmarkdown (Allaire et al. 2021; Xie, Allaire, and Grolemund 2018; Xie, Dervieux, and Riederer 2020), rmdformats (Barnier 2021), remedy (Fay, Sidi, and Smith 2018), knitr (Xie 2014, 2015, 2021) e citr (Aust 2019).
1. Instalação dos pacotes necessários
Antes de iniciarmos, é importante que você tenha os seguintes pacotes e dependências instalados:
#instala os pacotes, se for necessário:
pacotes = c("ggplot2","viridis","hrbrthemes","ggpubr")
for (p in setdiff(pacotes, installed.packages()[,"Package"])) {install.packages(p, dependencies=TRUE)}2. Checando e preparando os dados
Aqui, vamos usar como exemplo o Iris Dataset (Anderson 1935), que consiste em um data.frame com 150 observações (linhas) e 5 variáveis (colunas). Este famoso conjunto de dados fornece as medidas em centímetros das variáveis denominadas Sepal.Length, Sepal.Width, Petal.Length, Petal.Width e Species, respectivamente, para 50 flores de cada espécie de Iris (I. setosa, I. versicolor e I. virginica).
data("iris") #carrega o iris dataset
head(iris) #checa o topo dos dados## Sepal.Length Sepal.Width Petal.Length Petal.Width Species
## 1 5.1 3.5 1.4 0.2 setosa
## 2 4.9 3.0 1.4 0.2 setosa
## 3 4.7 3.2 1.3 0.2 setosa
## 4 4.6 3.1 1.5 0.2 setosa
## 5 5.0 3.6 1.4 0.2 setosa
## 6 5.4 3.9 1.7 0.4 setosastr(iris) #checa a estrutura dos dados## 'data.frame': 150 obs. of 5 variables:
## $ Sepal.Length: num 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ...
## $ Sepal.Width : num 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ...
## $ Petal.Length: num 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ...
## $ Petal.Width : num 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ...
## $ Species : Factor w/ 3 levels "setosa","versicolor",..: 1 1 1 1 1 1 1 1 1 1 ...summary(iris) #resume os dados## Sepal.Length Sepal.Width Petal.Length Petal.Width
## Min. :4.300 Min. :2.000 Min. :1.000 Min. :0.100
## 1st Qu.:5.100 1st Qu.:2.800 1st Qu.:1.600 1st Qu.:0.300
## Median :5.800 Median :3.000 Median :4.350 Median :1.300
## Mean :5.843 Mean :3.057 Mean :3.758 Mean :1.199
## 3rd Qu.:6.400 3rd Qu.:3.300 3rd Qu.:5.100 3rd Qu.:1.800
## Max. :7.900 Max. :4.400 Max. :6.900 Max. :2.500
## Species
## setosa :50
## versicolor:50
## virginica :50
##
##
## #cria subconjuntos para cada variável
CompSepal = data.frame(Especies=iris$Species, CompSepal=iris$Sepal.Length)
LargSepal = data.frame(Especies=iris$Species, LargSepal=iris$Sepal.Width)
CompPetal = data.frame(Especies=iris$Species, CompPetal=iris$Petal.Length)
LargPetal = data.frame(Especies=iris$Species, LargPetal=iris$Petal.Width)
#cria uma lista contendo os subconjuntos de dados
listaVariaveis = list(CompSepal=CompSepal,
LargSepal=LargSepal,
CompPetal=CompPetal,
LargPetal=LargPetal)
#cria um objeto que contenha os nomes das variáveis para usar no eixo Y de cada plot
nomesVariaveis = c("Comprimento da sépala (cm)",
"Largura da sépala (cm)",
"Comprimento da pétala (cm)",
"Largura da pétala (cm)")3. Loop for para gerar os boxplots
library("ggplot2") #para plotar os gráficos
library("viridis") #paleta de cores
library("hrbrthemes") #theme_ipsum()
#cria um objeto do tipo vetor para armazenar os resultados gerados
resultadoPlots = vector(mode="list", length=length(listaVariaveis))
#gera o plot de cada variável (em subconjunto) dentro de um loop
for(i in 1:length(listaVariaveis)){
resultadoPlots[[i]] = ggplot(listaVariaveis[[i]],
#veja também aes() em vez de aes_string()
aes_string(x=names(listaVariaveis[[i]][1]), #pega a coluna que corresponde aos nomes das spp em cada subconjunto
y=names(listaVariaveis[[i]][2]), #pega a coluna que corresponde aos nomes das variáveis em cada subconjunto
fill=names(listaVariaveis[[i]][1]))) + #define as cores por spp
geom_boxplot() +
scale_fill_viridis(discrete=TRUE, alpha=0.6) +
geom_jitter(color="black", size=0.4, alpha=0.9) +
theme_ipsum() +
theme(legend.position="none",
axis.title.x=element_text(size=12),
axis.title.y=element_text(size=12),
axis.text=element_text(size=10),
axis.text.x=element_text(face="italic")) + #para que os nomes no eixo X fiquem em itálico
xlab("Iris spp.") +
ylab(nomesVariaveis[i])
print(resultadoPlots[[i]]) #para ver o resultado
}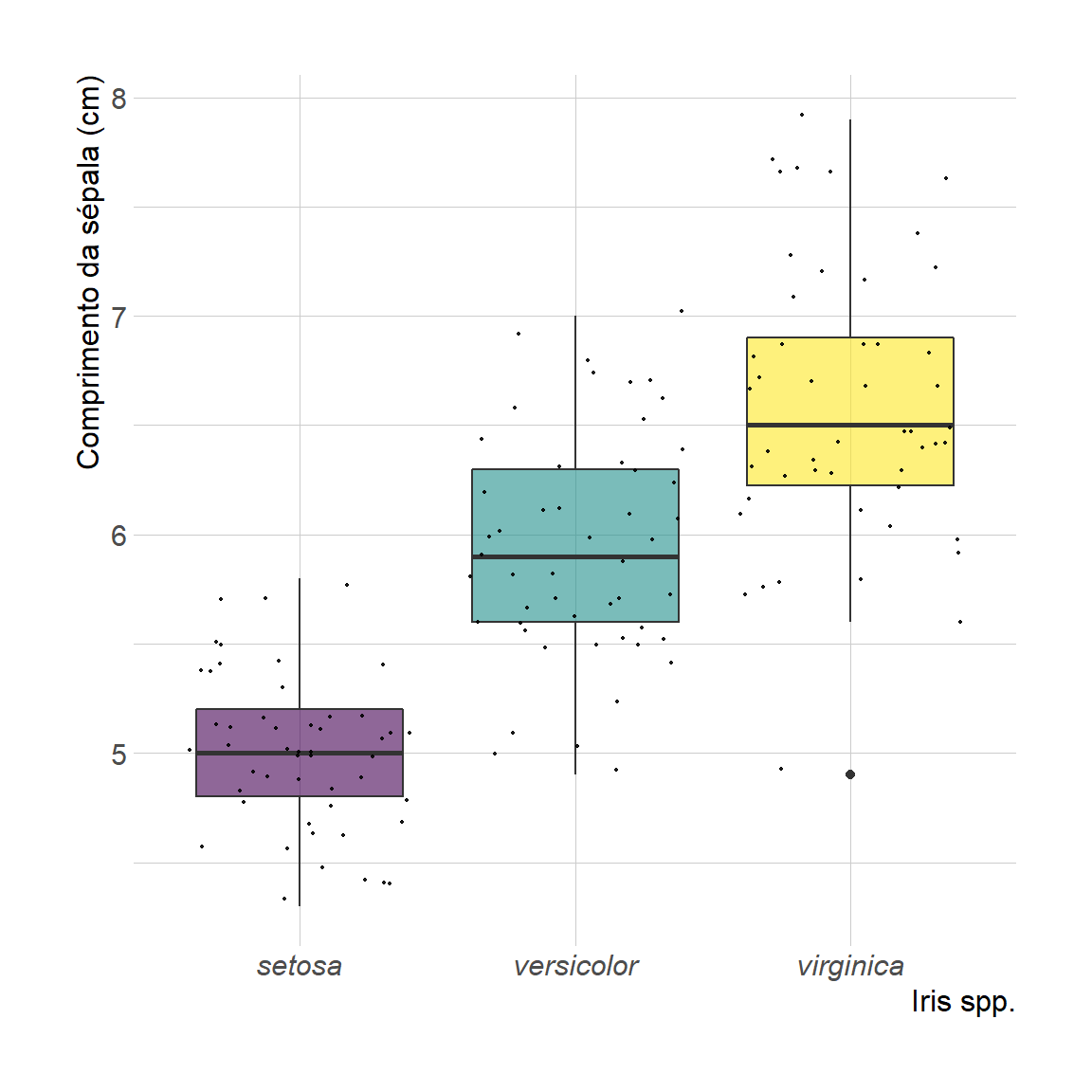
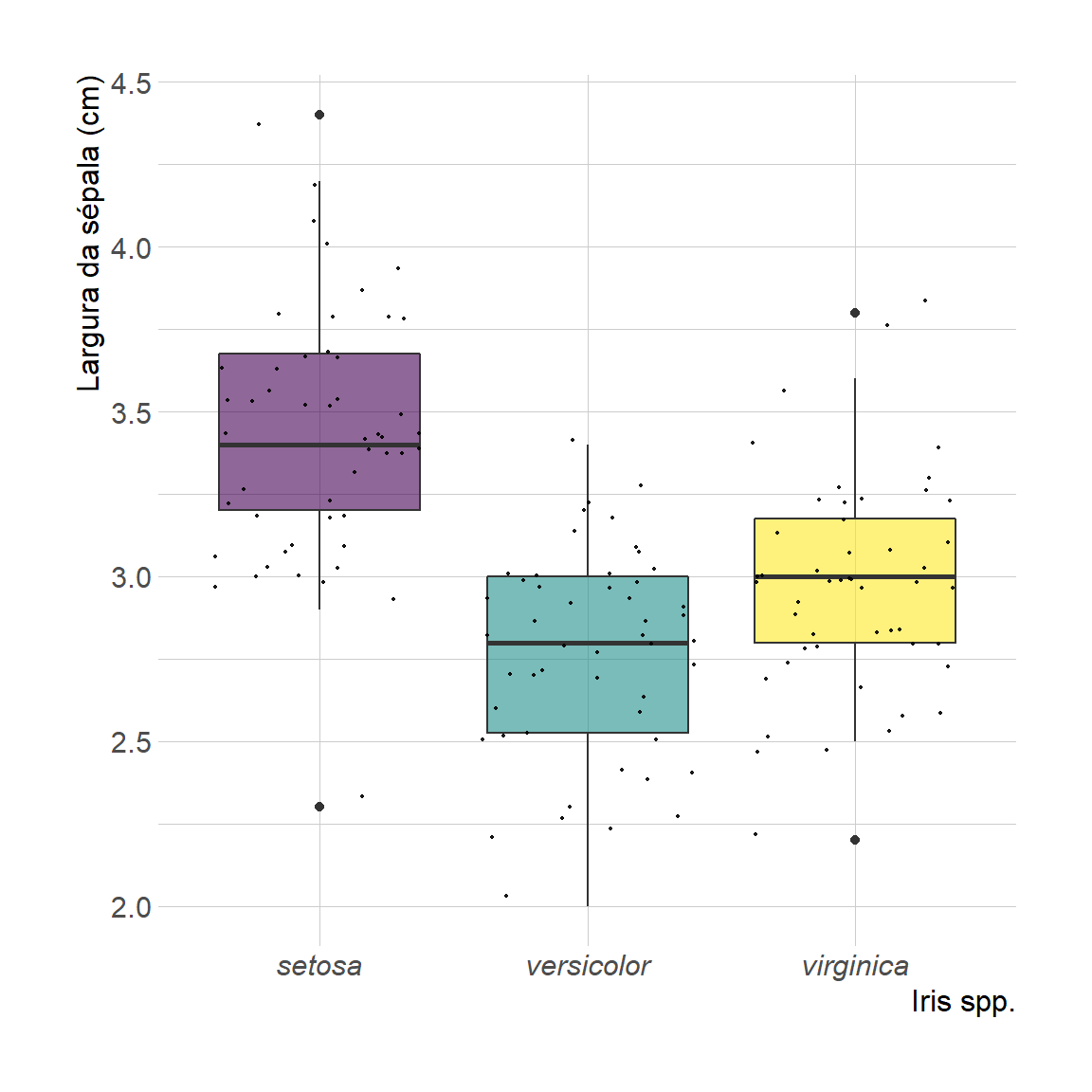
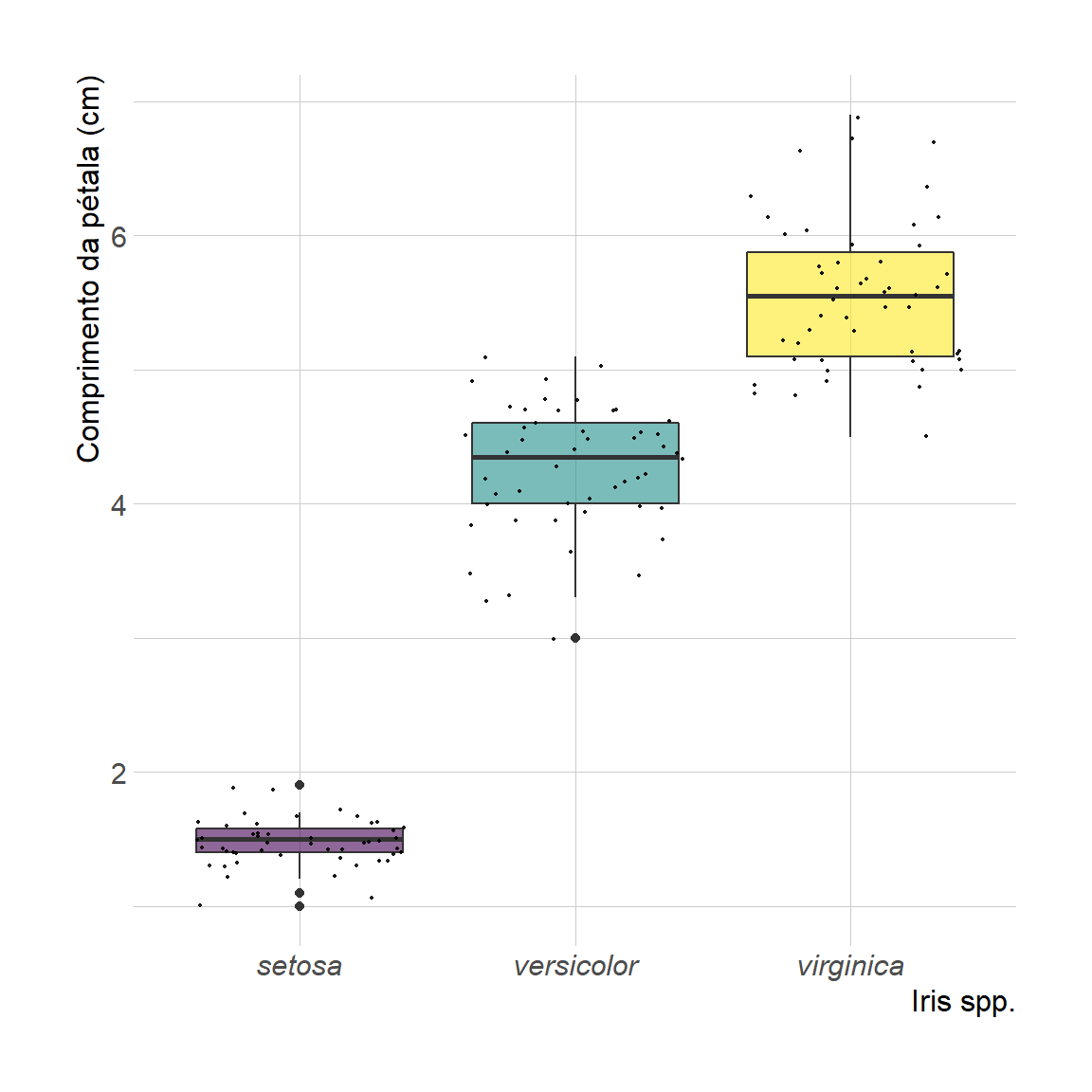
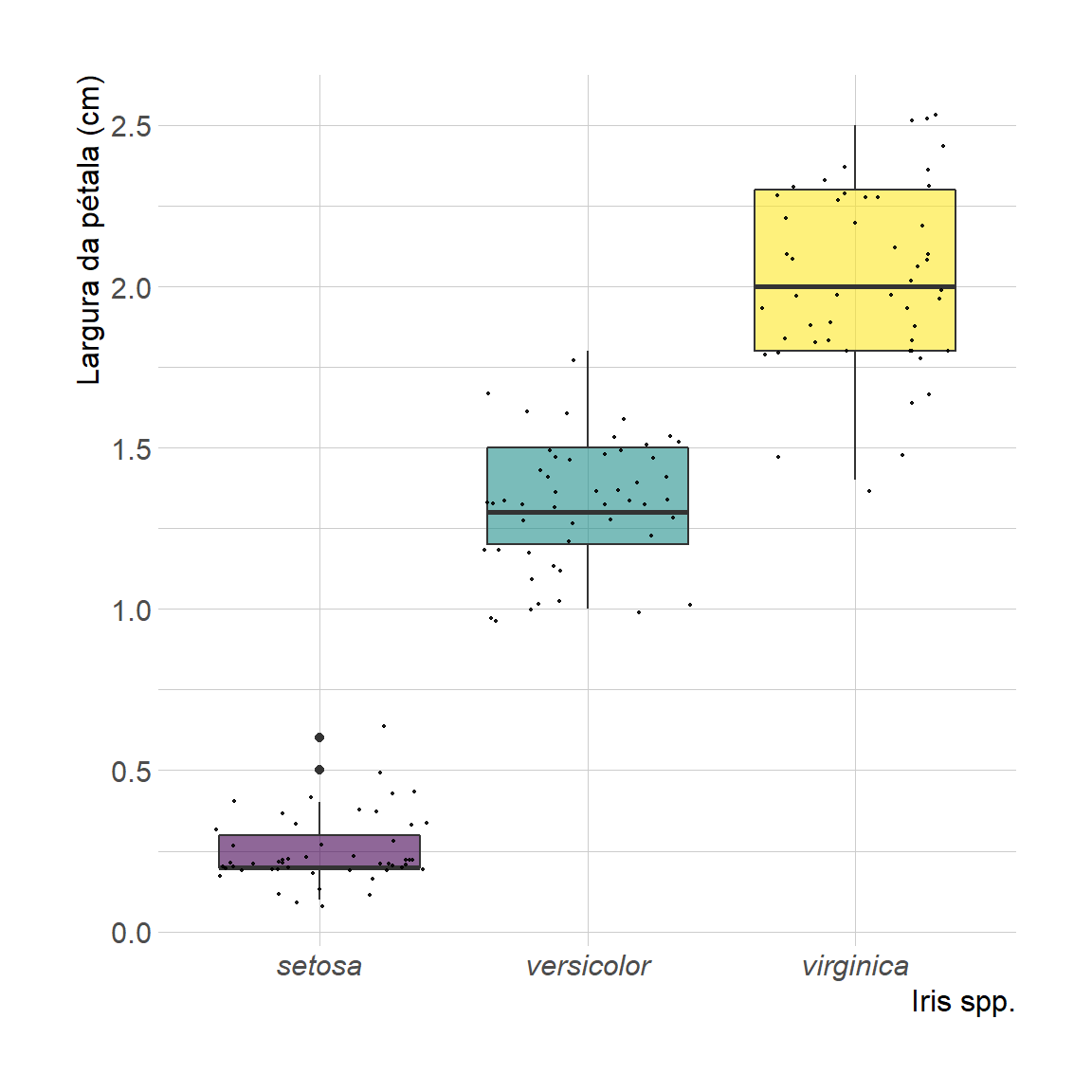
4. Plota os boxplots em uma única figura
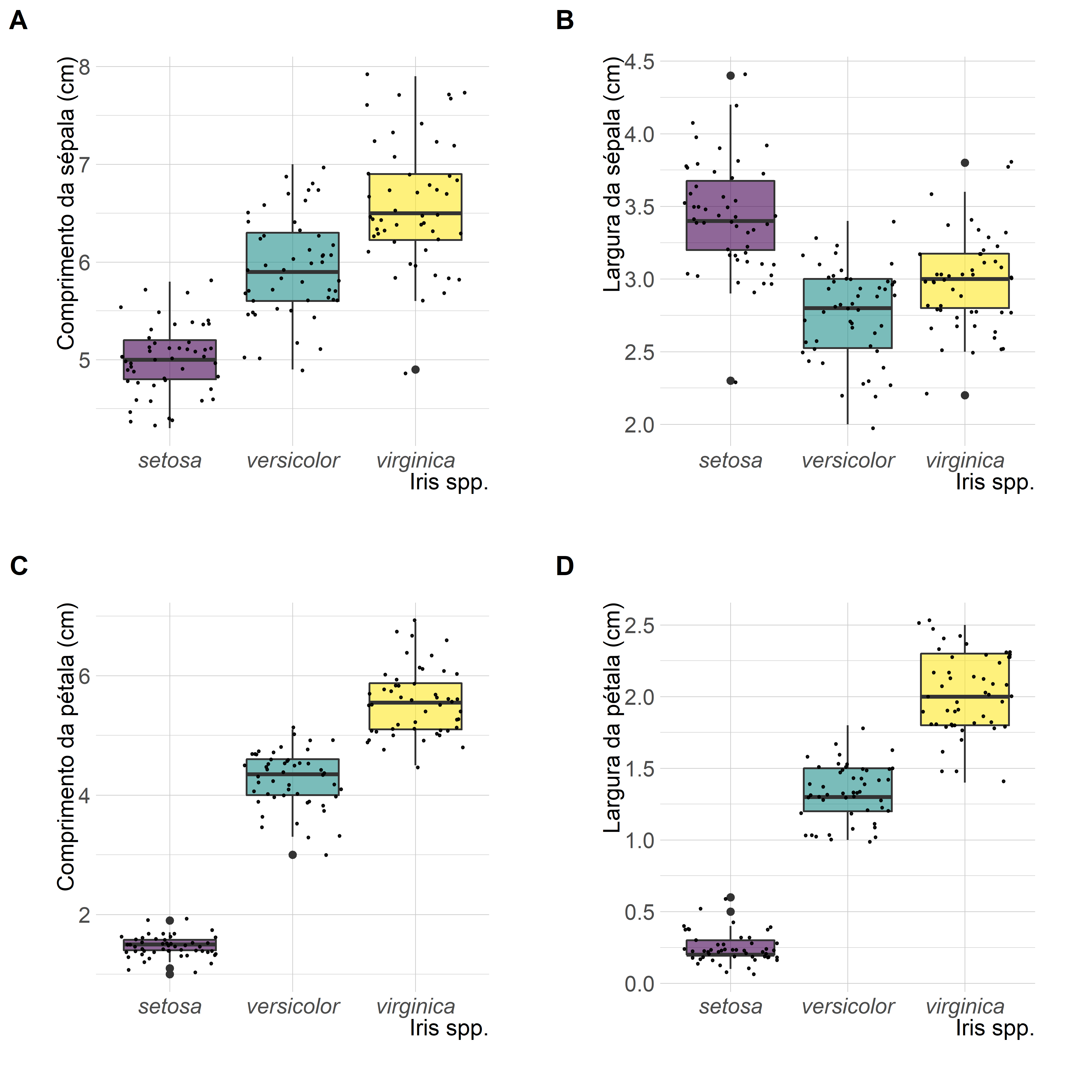
5. Salva a figura em um arquivo .tiff de alta qualidade
ggsave("FiguraBoxplots.tiff", plot=figuraBoxplots,
width=22, height=20, units="cm", dpi=300, compression="lzw") #define os parâmetros da imagemPostagem originalmente publicada no meu perfil do RPubs. Toda semana tem conteúdo novo por lá!
Até a próxima! See you soon!
Referências
Allaire, JJ, Yihui Xie, Jonathan McPherson, Javier Luraschi, Kevin Ushey, Aron Atkins, Hadley Wickham, Joe Cheng, Winston Chang, and Richard Iannone. 2021. Rmarkdown: Dynamic Documents for R. https://github.com/rstudio/rmarkdown.
Anderson, Edgar. 1935. “The Irises of the Gaspe Peninsula.” Bulletin of the American Iris Society 59: 2–5.
Aust, Frederik. 2019. Citr: ’RStudio’ Add-in to Insert Markdown Citations. https://github.com/crsh/citr.
Barnier, Julien. 2021. Rmdformats: HTML Output Formats and Templates for ’Rmarkdown’ Documents. https://CRAN.R-project.org/package=rmdformats.
Fay, Colin, Jonathan Sidi, and Luke Smith. 2018. Remedy: ’RStudio’ Addins to Simplify ’Markdown’ Writing. https://CRAN.R-project.org/package=remedy.
Garnier, Simon. 2018. Viridis: Default Color Maps from ’Matplotlib’. https://CRAN.R-project.org/package=viridis.
Kassambara, Alboukadel. 2020. Ggpubr: ’Ggplot2’ Based Publication Ready Plots. https://CRAN.R-project.org/package=ggpubr.
Rudis, Bob. 2020. Hrbrthemes: Additional Themes, Theme Components and Utilities for ’Ggplot2’. https://CRAN.R-project.org/package=hrbrthemes.
Wickham, Hadley. 2016. Ggplot2: Elegant Graphics for Data Analysis. Springer-Verlag New York. https://ggplot2.tidyverse.org.
Xie, Yihui. 2014. “Knitr: A Comprehensive Tool for Reproducible Research in R.” In Implementing Reproducible Computational Research, edited by Victoria Stodden, Friedrich Leisch, and Roger D. Peng. Chapman; Hall/CRC. http://www.crcpress.com/product/isbn/9781466561595.
———. 2015. Dynamic Documents with R and Knitr. 2nd ed. Boca Raton, Florida: Chapman; Hall/CRC. https://yihui.org/knitr/.
———. 2021. Knitr: A General-Purpose Package for Dynamic Report Generation in R. https://yihui.org/knitr/.
Xie, Yihui, J. J. Allaire, and Garrett Grolemund. 2018. R Markdown: The Definitive Guide. Boca Raton, Florida: Chapman; Hall/CRC. https://bookdown.org/yihui/rmarkdown.
Xie, Yihui, Christophe Dervieux, and Emily Riederer. 2020. R Markdown Cookbook. Boca Raton, Florida: Chapman; Hall/CRC. https://bookdown.org/yihui/rmarkdown-cookbook.