Criando um mapa de distribuição no R [Portuguese text]
Aprenda a obter dados de ocorrência do GBIF, a filtrar e a processar esses dados para gerar um mapa simples de distribuição geográfica.
Neste tutorial você aprenderá a obter dados de ocorrência do GBIF - Global Biodiversity Information Facility, a filtrar e a processar esses dados para gerar um mapa simples de distribuição geográfica.
Esta postagem foi inteiramente produzida em ambiente R Studio, usando os pacotes rmarkdown (Allaire et al. 2021; Xie, Allaire, and Grolemund 2018; Xie, Dervieux, and Riederer 2020), rmdformats (Barnier 2021), remedy (Fay, Sidi, and Smith 2018), knitr (Xie 2014, 2015, 2021) e citr (Aust 2019).
1. Instalação dos pacotes necessários
Antes de iniciarmos, é importante que você tenha os seguintes pacotes e dependências instalados:
rgbif(Chamberlain and Boettiger 2017; Chamberlain et al. 2021)dplyr(Wickham et al. 2020)tidyr(Wickham 2020)CoordinateCleaner(Zizka et al. 2019)maps(Richard A. Becker, Ray Brownrigg. Enhancements by Thomas P Minka, and Deckmyn. 2018)prettymapr(Dunnington 2017)GISTools(Brunsdon and Chen 2014)readr(Wickham and Hester 2020)
#instala os pacotes, se for necessário:
pacotes = c("rgbif","dplyr","tidyr","CoordinateCleaner","maps","prettymapr","GISTools","readr")
for (p in setdiff(pacotes, installed.packages()[,"Package"])) {install.packages(p, dependencies=TRUE)}2. Obtenção, filtragem e limpeza dos dados de ocorrência do GBIF
library("rgbif")
#retorna apenas as ocorrências que possuem coordenadas geográficas
#no argumento 'scientificName' você também pode definir outros níveis taxonômicos (p.ex. espécie, família)
occEcclinusa = occ_data(scientificName="Ecclinusa", hasCoordinate=TRUE)
#mostra as 10 primeiras linhas e 5 primeiras colunas dos dados
occEcclinusa$data[1:10,1:5]## # A tibble: 10 x 5
## key scientificName decimalLatitude decimalLongitude issues
## <chr> <chr> <dbl> <dbl> <chr>
## 1 201390~ Ecclinusa Mart. -22.5 -43.0 cdround,gass84,i~
## 2 286919~ Ecclinusa Mart. -7.06 -76.0 cdround,gass84,o~
## 3 286920~ Ecclinusa Mart. -7.05 -76.0 cdround,gass84,o~
## 4 301342~ Ecclinusa ramiflo~ -25.3 -48.3 gass84,diffown
## 5 183573~ Ecclinusa ramiflo~ -22.4 -43.7 cdround,gass84,i~
## 6 183573~ Ecclinusa ramiflo~ -22.4 -43.7 cdround,gass84,i~
## 7 225252~ Ecclinusa ramiflo~ -25.2 -48.3 cdround,gass84,r~
## 8 251269~ Ecclinusa ramiflo~ -25.2 -48.3 gass84,diffown
## 9 299686~ Ecclinusa ramiflo~ -25.2 -48.3 cdround,gass84,r~
## 10 199382~ Ecclinusa guianen~ -18.8 -40.4 gass84library("dplyr")
#filtra apenas as colunas de interesse
filtroColunas = occEcclinusa$data %>% dplyr::select(scientificName, decimalLatitude, decimalLongitude)
#mostra o topo dos dados
head(filtroColunas)## # A tibble: 6 x 3
## scientificName decimalLatitude decimalLongitude
## <chr> <dbl> <dbl>
## 1 Ecclinusa Mart. -22.5 -43.0
## 2 Ecclinusa Mart. -7.06 -76.0
## 3 Ecclinusa Mart. -7.05 -76.0
## 4 Ecclinusa ramiflora Mart. -25.3 -48.3
## 5 Ecclinusa ramiflora Mart. -22.4 -43.7
## 6 Ecclinusa ramiflora Mart. -22.4 -43.7#quantas linhas e colunas?
dim(filtroColunas)## [1] 500 3#quantos registros por spp?
table(filtroColunas$scientificName)##
## Chrysophyllum guianense (Eyma) Baehni
## 6
## Ecclimusa Mart. ex A.DC.
## 1
## Ecclinusa atabapoensis (Aubrév.) T.D.Penn.
## 25
## Ecclinusa bullata T.D.Penn.
## 6
## Ecclinusa guianensis Eyma
## 115
## Ecclinusa lanceolata (Mart. & Eichler) Pierre
## 90
## Ecclinusa Mart.
## 67
## Ecclinusa parviflora T.D.Penn.
## 1
## Ecclinusa ramiflora Mart.
## 189#filtrando as spp - retorna os nomes únicos
quaisSpp = unique(filtroColunas$scientificName); quaisSpp## [1] "Ecclinusa Mart."
## [2] "Ecclinusa ramiflora Mart."
## [3] "Ecclinusa guianensis Eyma"
## [4] "Ecclinusa parviflora T.D.Penn."
## [5] "Ecclinusa lanceolata (Mart. & Eichler) Pierre"
## [6] "Ecclinusa atabapoensis (Aubrév.) T.D.Penn."
## [7] "Chrysophyllum guianense (Eyma) Baehni"
## [8] "Ecclinusa bullata T.D.Penn."
## [9] "Ecclimusa Mart. ex A.DC."#selecionando apenas os nomes válidos
selecionaSpp = quaisSpp[c(2:6,8)]
filtroSpp = filtroColunas[filtroColunas$scientificName %in% selecionaSpp, ]
head(filtroSpp)## # A tibble: 6 x 3
## scientificName decimalLatitude decimalLongitude
## <chr> <dbl> <dbl>
## 1 Ecclinusa ramiflora Mart. -25.3 -48.3
## 2 Ecclinusa ramiflora Mart. -22.4 -43.7
## 3 Ecclinusa ramiflora Mart. -22.4 -43.7
## 4 Ecclinusa ramiflora Mart. -25.2 -48.3
## 5 Ecclinusa ramiflora Mart. -25.2 -48.3
## 6 Ecclinusa ramiflora Mart. -25.2 -48.3#busca os pontos problemáticos conforme os parâmetros definidos - pode demorar um pouquinho!
limpaCoordenadas = CoordinateCleaner::clean_coordinates(
x = filtroSpp,
species = "scientificName",
lon = "decimalLongitude",
lat = "decimalLatitude",
tests = c("duplicates", #duplicatas
"equal", #coordenadas iguais
"seas", #pontos no mar
"zeros" #zeros e pontos em que lat = lon
)
)## OGR data source with driver: ESRI Shapefile
## Source: "C:\Users\cc_va\AppData\Local\Temp\RtmpKcWVmT", layer: "ne_50m_land"
## with 1420 features
## It has 3 fields
## Integer64 fields read as strings: scalerank#resultado da marcação de pontos problemáticos
limpaCoordenadas %>% head## scientificName decimalLatitude decimalLongitude .val .equ .zer
## 1 Ecclinusa ramiflora Mart. -25.28083 -48.31333 TRUE TRUE TRUE
## 2 Ecclinusa ramiflora Mart. -22.35694 -43.73500 TRUE TRUE TRUE
## 3 Ecclinusa ramiflora Mart. -22.35694 -43.73472 TRUE TRUE TRUE
## 4 Ecclinusa ramiflora Mart. -25.17111 -48.30139 TRUE TRUE TRUE
## 5 Ecclinusa ramiflora Mart. -25.17111 -48.30139 TRUE TRUE TRUE
## 6 Ecclinusa ramiflora Mart. -25.17111 -48.30139 TRUE TRUE TRUE
## .sea .dpl .summary
## 1 TRUE TRUE TRUE
## 2 TRUE TRUE TRUE
## 3 TRUE TRUE TRUE
## 4 TRUE TRUE TRUE
## 5 TRUE FALSE FALSE
## 6 TRUE FALSE FALSEsummary(limpaCoordenadas)## .val .equ .zer .sea .dpl .summary
## 0 0 0 10 131 140#para excluir os pontos marcados como problemáticos
#TRUE = coordenadas limpas
#FALSE = coordenadas potencialmente problemáticas
filtroSpp_limpo = filtroSpp %>% dplyr::filter(limpaCoordenadas$.summary==TRUE)
filtroSpp_limpo## # A tibble: 286 x 3
## scientificName decimalLatitude decimalLongitude
## <chr> <dbl> <dbl>
## 1 Ecclinusa ramiflora Mart. -25.3 -48.3
## 2 Ecclinusa ramiflora Mart. -22.4 -43.7
## 3 Ecclinusa ramiflora Mart. -22.4 -43.7
## 4 Ecclinusa ramiflora Mart. -25.2 -48.3
## 5 Ecclinusa guianensis Eyma -18.8 -40.4
## 6 Ecclinusa ramiflora Mart. 0.0126 -71.0
## 7 Ecclinusa ramiflora Mart. -15.6 -39.4
## 8 Ecclinusa ramiflora Mart. 1.12 -72.8
## 9 Ecclinusa guianensis Eyma 1.36 -72.9
## 10 Ecclinusa guianensis Eyma 2.69 -57.1
## # ... with 276 more rows#compara os dados de ocorrência por spp (antes, durante e depois do tratamento)
table(filtroColunas$scientificName)##
## Chrysophyllum guianense (Eyma) Baehni
## 6
## Ecclimusa Mart. ex A.DC.
## 1
## Ecclinusa atabapoensis (Aubrév.) T.D.Penn.
## 25
## Ecclinusa bullata T.D.Penn.
## 6
## Ecclinusa guianensis Eyma
## 115
## Ecclinusa lanceolata (Mart. & Eichler) Pierre
## 90
## Ecclinusa Mart.
## 67
## Ecclinusa parviflora T.D.Penn.
## 1
## Ecclinusa ramiflora Mart.
## 189table(filtroSpp$scientificName)##
## Ecclinusa atabapoensis (Aubrév.) T.D.Penn.
## 25
## Ecclinusa bullata T.D.Penn.
## 6
## Ecclinusa guianensis Eyma
## 115
## Ecclinusa lanceolata (Mart. & Eichler) Pierre
## 90
## Ecclinusa parviflora T.D.Penn.
## 1
## Ecclinusa ramiflora Mart.
## 189table(filtroSpp_limpo$scientificName)##
## Ecclinusa atabapoensis (Aubrév.) T.D.Penn.
## 17
## Ecclinusa bullata T.D.Penn.
## 5
## Ecclinusa guianensis Eyma
## 65
## Ecclinusa lanceolata (Mart. & Eichler) Pierre
## 65
## Ecclinusa parviflora T.D.Penn.
## 1
## Ecclinusa ramiflora Mart.
## 1333. Exporta os dados de ocorrência processados
#ordena a planilha a partir do nome das spp
geodataEcclinusa = filtroSpp_limpo %>% arrange(desc(scientificName))
#exporta a planilha em .csv
readr::write_csv(geodataEcclinusa, paste0("OcorrenciasGBIF_Ecclinusa.csv"))4. Prepara os dados para gerar o mapa
#importa o arquivo contendo os dados espaciais
#geodataEcclinusa = readr::read_csv("OcorrenciasGBIF_Ecclinusa.csv")
#define os limites para o mapa
xlim = range(geodataEcclinusa$decimalLongitude, na.rm=TRUE) + c(-6,6)
ylim = range(geodataEcclinusa$decimalLatitude, na.rm=TRUE) + c(-6,6)
#filtro contendo as coordenadas geográficas de cada sp
ramiflora = geodataEcclinusa[geodataEcclinusa$scientificName %in% selecionaSpp[1], 3:2] #3:2 para inverter a ordem para lon e lat
guianensis = geodataEcclinusa[geodataEcclinusa$scientificName %in% selecionaSpp[2], 3:2]
parviflora = geodataEcclinusa[geodataEcclinusa$scientificName %in% selecionaSpp[3], 3:2]
lanceolata = geodataEcclinusa[geodataEcclinusa$scientificName %in% selecionaSpp[4], 3:2]
atabapoensis = geodataEcclinusa[geodataEcclinusa$scientificName %in% selecionaSpp[5], 3:2]
bullata = geodataEcclinusa[geodataEcclinusa$scientificName %in% selecionaSpp[6], 3:2]
#nomes das spp personalizados para a legenda - nomes científicos em itálico
nomeSpp_legenda =c(expression(paste(italic("Ecclinusa ramiflora "), "Mart.")),
expression(paste(italic("Ecclinusa guianensis "), "Eyma")),
expression(paste(italic("Ecclinusa parviflora "), "T.D.Penn.")),
expression(paste(italic("Ecclinusa lanceolata "), "(Mart. & Eichler) Pierre")),
expression(paste(italic("Ecclinusa atabapoensis "), "(Aubrév.) T.D.Penn.")),
expression(paste(italic("Ecclinusa bullata "), "T.D.Penn."))
)
#cria uma paleta de cores para representar cada sp
paletaCustomizada = c("#0072B2","#882255","#BBCC33","#EE6677","#d55E00","#117733") 5. Plota o mapa
library(maps)
#plota as camadas
map("world", "Brazil", fill=T, col="grey90", xlim=xlim, ylim=ylim)
map(,,add=T,xlim=xlim, ylim=ylim)
#plota o conjunto de pontos por espécie
points(ramiflora, pch=19, cex=1, col=paletaCustomizada[1])
points(guianensis, pch=19, cex=1, col=paletaCustomizada[2])
points(parviflora, pch=19, cex=1, col=paletaCustomizada[3])
points(lanceolata, pch=19, cex=1, col=paletaCustomizada[4])
points(atabapoensis, pch=19, cex=1, col=paletaCustomizada[5])
points(bullata, pch=19, cex=1, col=paletaCustomizada[6])
#plota alguns elementos de mapa
abline(h=0, lwd=0.8, lty=2, col="gray56") #adiciona uma linha para representar a do equador (opcional)
#grid(lty=2, lwd=0.8, col="gray56") #adiciona um grid (opcional) - caso opte pelo grid seria esteticamente melhor não executar o comando acima
prettymapr::addnortharrow("bottomleft", scale=0.7) #adiciona o símbolo do Norte
#GISTools::north.arrow(xb=-85, yb=-29,len=0.8, lab="N") #outra opção de símbolo do Norte
maps::map.scale(x=-42,y=-28, ratio=FALSE, cex=0.8) #adiciona escala; em x e y você também pode alterar a posição
map.axes() #delimita a figura
#plota a legenda
legend(x=-53, y=16, #aqui você pode alterar a posição da legenda
legend=nomeSpp_legenda, #nomes das espécies
col=paletaCustomizada, #cores definidas anteriormente
pch=19,
pt.cex=1, inset=0.05, cex=0.8, bty="n") #outros parâmetros da legenda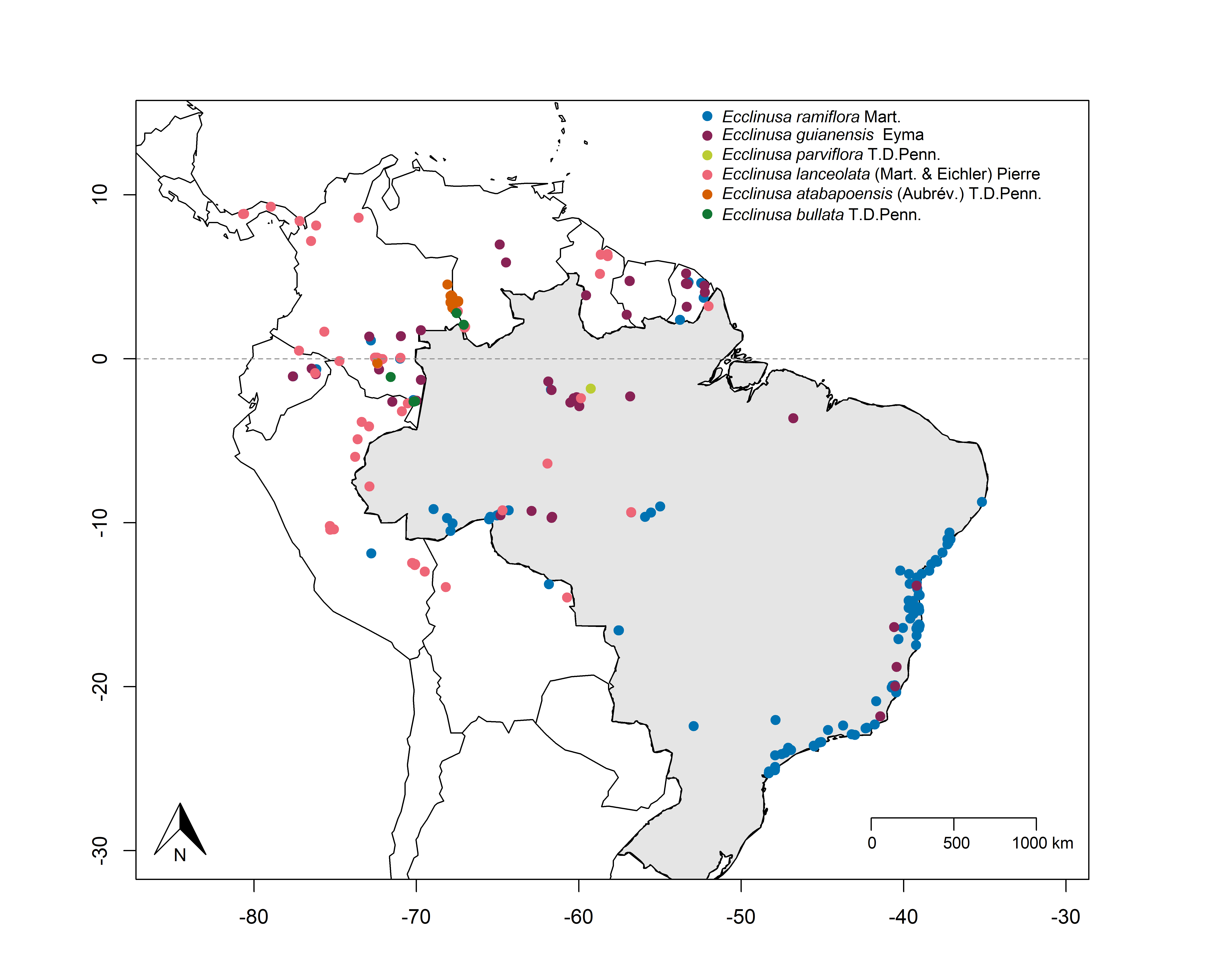
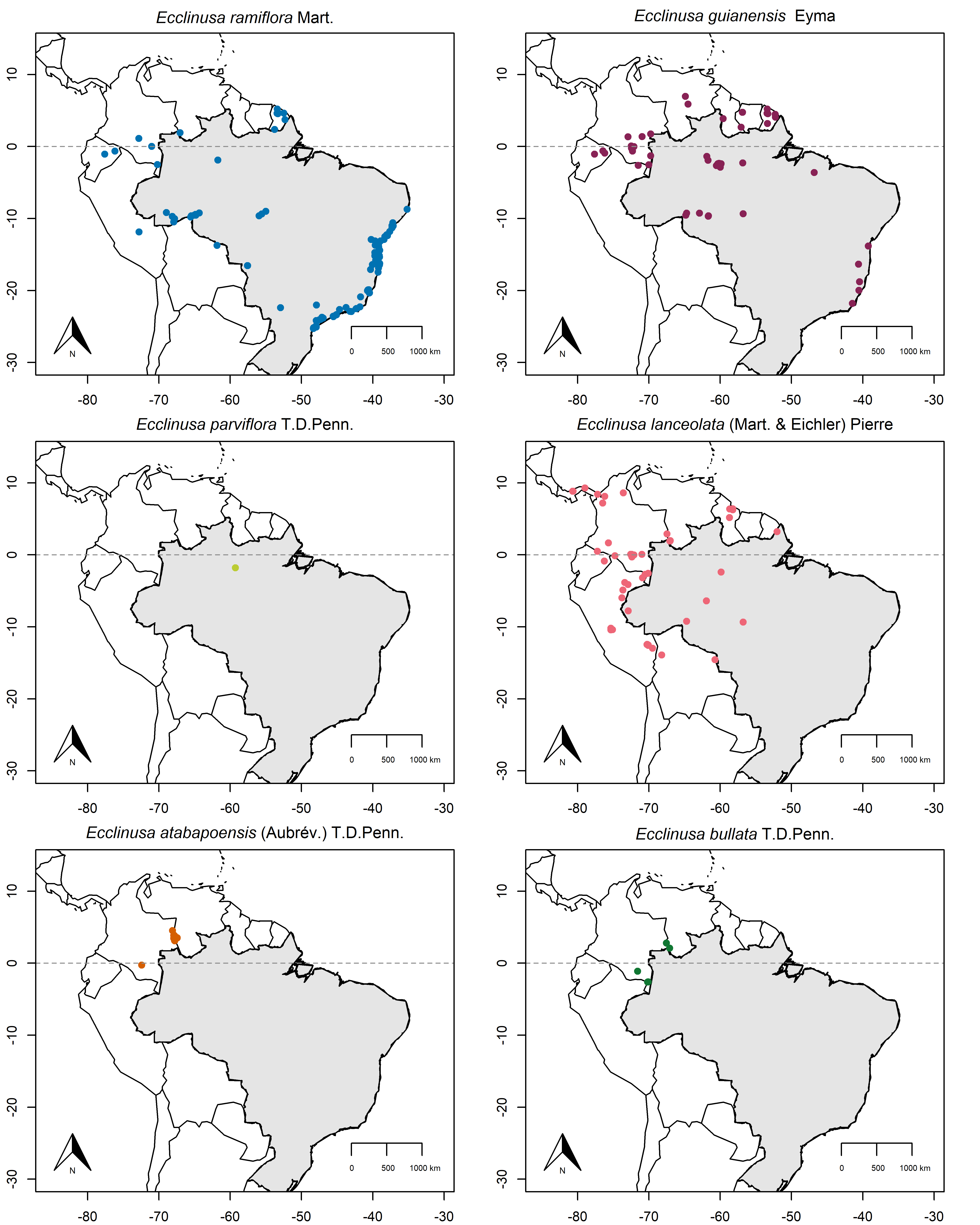
Caso queira evitar sobreposição de pontos, você pode plotar a distribuição das espécies separadamente usando o código abaixo:
#cria uma lista com os filtros que criamos anteriormente contendo as coordenadas de cada sp
listaSpp = list(ramiflora=ramiflora,
guianensis=guianensis,
parviflora=parviflora,
lanceolata=lanceolata,
atabapoensis=atabapoensis,
bullata=bullata)
#define alguns parâmetros gráficos (linhas, colunas, margem)
par(mfrow=c(3,2), mar=c(0,0,0,0))
#plota os mapas de cada sp dentro de um loop
for(i in 1:length(listaSpp)){
map("world", "Brazil", fill=T, col="grey90", xlim=xlim, ylim=ylim)
title(nomeSpp_legenda[i], line=1) #optei por não plotar a legenda e sim o nome das spp no título de cada plot
map(,,add=T,xlim=xlim, ylim=ylim)
points(listaSpp[[i]], pch=19, cex=1, col=paletaCustomizada[i])
abline(h=0, lwd=0.8, lty=2, col="gray56")
prettymapr::addnortharrow("bottomleft", scale=0.5) #altere os argumentos que envolvem posição e tamanho conforme sua necessidade
maps::map.scale(x=-43,y=-25, ratio=FALSE, cex=0.6)
map.axes()
}
6. Salva o plot em um arquivo tiff
#OPÇÃO 1
#abre um dispositivo .tiff para salvar a figura
tiff("MapaDistribuicaoEcclinusaTodasSpp.tiff",width=20, height=20, units="cm", comp="lzw", res=300)
library(maps)
map("world", "Brazil", fill=T, col="grey90", xlim=xlim, ylim=ylim)
map(,,add=T,xlim=xlim, ylim=ylim)
points(ramiflora, pch=19, cex=1, col=paletaCustomizada[1])
points(guianensis, pch=19, cex=1, col=paletaCustomizada[2])
points(parviflora, pch=19, cex=1, col=paletaCustomizada[3])
points(lanceolata, pch=19, cex=1, col=paletaCustomizada[4])
points(atabapoensis, pch=19, cex=1, col=paletaCustomizada[5])
points(bullata, pch=19, cex=1, col=paletaCustomizada[6])
abline(h=0, lwd=0.8, lty=2, col="gray56")
prettymapr::addnortharrow("bottomleft", scale=0.7)
maps::map.scale(x=-42,y=-28, ratio=FALSE, cex=0.8)
map.axes()
legend(x=-53, y=16,
legend=nomeSpp_legenda,
col=paletaCustomizada,
pch=19,
pt.cex=1, inset=0.05, cex=0.8, bty="n")
#fecha o dispositivo tiff e salva a figura
dev.off()## png
## 2#OPÇÃO 2
tiff("MapaDistribuicaoEcclinusaPorSpp.tiff",width=20, height=20, units="cm", comp="lzw", res=300)
par(mfrow=c(3,2), mar=c(0,0,0,0))
for(i in 1:length(listaSpp)){
map("world", "Brazil", fill=T, col="grey90", xlim=xlim, ylim=ylim)
title(nomeSpp_legenda[i], line=1)
map(,,add=T,xlim=xlim, ylim=ylim)
points(listaSpp[[i]], pch=19, cex=1, col=paletaCustomizada[i])
abline(h=0, lwd=0.8, lty=2, col="gray56")
prettymapr::addnortharrow("bottomleft", scale=0.5)
maps::map.scale(x=-43,y=-25, ratio=FALSE, cex=0.6)
map.axes()
}
dev.off()## png
## 27. Uso dos dados do GBIF
Como boa prática científica, é importante lembrar que o contrato do usuário que rege o uso de dados do GBIF exige especificamente que todos os usuários devem reconhecer publicamente, seguindo a convenção científica de citar fontes em conjunto com o uso dos dados. Saiba mais sobre isso clicando aqui.
Através do pacote rgbif você pode usar os seguintes comandos para obter essas informações:
x = occ_search(scientificName="Ecclinusa", limit=10)
gbif_citation(x)## [[1]]
## <<rgbif citation>>
## Citation: Forzza R, Estevão L A, Dalcin E (2021). RB - Rio de Janeiro
## Botanical Garden Herbarium Collection. Version 84.223. Instituto de
## Pesquisas Jardim Botanico do Rio de Janeiro. Occurrence dataset
## https://doi.org/10.15468/7ep9i2 accessed via GBIF.org on 2021-07-02..
## Accessed from R via rgbif (https://github.com/ropensci/rgbif) on
## 2021-07-02
## Rights: http://creativecommons.org/licenses/by/4.0/legalcode
##
## [[2]]
## <<rgbif citation>>
## Citation: Ferreira Dutra V (2021). VIES - Herbário Central da Universidade
## Federal do Espírito Santo. Version 1.79. Universidade Federal do
## Espírito Santo. Occurrence dataset https://doi.org/10.15468/rt7ybs
## accessed via GBIF.org on 2021-07-02.. Accessed from R via rgbif
## (https://github.com/ropensci/rgbif) on 2021-07-02
## Rights: http://creativecommons.org/licenses/by-nc/4.0/legalcode
##
## [[3]]
## <<rgbif citation>>
## Citation: Thomaz L D, Forzza R, Lanna J (2021). VIES herbarium -
## Universidade Federal do Espírito Santo - Herbário Virtual REFLORA.
## Version 1.250. Instituto de Pesquisas Jardim Botanico do Rio de
## Janeiro. Occurrence dataset https://doi.org/10.15468/kasze8 accessed
## via GBIF.org on 2021-07-02.. Accessed from R via rgbif
## (https://github.com/ropensci/rgbif) on 2021-07-02
## Rights: http://creativecommons.org/licenses/by/4.0/legalcode
##
## [[4]]
## <<rgbif citation>>
## Citation: Solomon J, Stimmel H (2021). Tropicos Specimen Data. Missouri
## Botanical Garden. Occurrence dataset https://doi.org/10.15468/hja69f
## accessed via GBIF.org on 2021-07-02.. Accessed from R via rgbif
## (https://github.com/ropensci/rgbif) on 2021-07-02
## Rights: http://creativecommons.org/licenses/by/4.0/legalcode
##
## [[5]]
## <<rgbif citation>>
## Citation: Leandro Brotto M (2021). MBM - Herbário do Museu Botânico
## Municipal. Version 1.78. Museu Botânico Municipal. Occurrence dataset
## https://doi.org/10.15468/g6ppmt accessed via GBIF.org on 2021-07-02..
## Accessed from R via rgbif (https://github.com/ropensci/rgbif) on
## 2021-07-02
## Rights: http://creativecommons.org/licenses/by-nc/4.0/legalcodePostagem originalmente publicada no meu perfil do RPubs. Toda semana tem conteúdo novo por lá!
Até a próxima! See you soon!
Referências
Allaire, JJ, Yihui Xie, Jonathan McPherson, Javier Luraschi, Kevin Ushey, Aron Atkins, Hadley Wickham, Joe Cheng, Winston Chang, and Richard Iannone. 2021. Rmarkdown: Dynamic Documents for R. https://github.com/rstudio/rmarkdown.
Aust, Frederik. 2019. Citr: ’RStudio’ Add-in to Insert Markdown Citations. https://github.com/crsh/citr.
Barnier, Julien. 2021. Rmdformats: HTML Output Formats and Templates for ’Rmarkdown’ Documents. https://CRAN.R-project.org/package=rmdformats.
Brunsdon, Chris, and Hongyan Chen. 2014. GISTools: Some Further Gis Capabilities for R. https://CRAN.R-project.org/package=GISTools.
Chamberlain, Scott, Vijay Barve, Dan Mcglinn, Damiano Oldoni, Peter Desmet, Laurens Geffert, and Karthik Ram. 2021. Rgbif: Interface to the Global Biodiversity Information Facility Api. https://CRAN.R-project.org/package=rgbif.
Chamberlain, Scott, and Carl Boettiger. 2017. “R Python, and Ruby Clients for Gbif Species Occurrence Data.” PeerJ PrePrints, no. 5: e3304v1. https://doi.org/10.7287/peerj.preprints.3304v1.
Dunnington, Dewey. 2017. Prettymapr: Scale Bar, North Arrow, and Pretty Margins in R. https://CRAN.R-project.org/package=prettymapr.
Fay, Colin, Jonathan Sidi, and Luke Smith. 2018. Remedy: ’RStudio’ Addins to Simplify ’Markdown’ Writing. https://CRAN.R-project.org/package=remedy.
Richard A. Becker, Original S code by, Allan R. Wilks. R version by Ray Brownrigg. Enhancements by Thomas P Minka, and Alex Deckmyn. 2018. Maps: Draw Geographical Maps. https://CRAN.R-project.org/package=maps.
Wickham, Hadley. 2020. Tidyr: Tidy Messy Data. https://CRAN.R-project.org/package=tidyr.
Wickham, Hadley, Romain François, Lionel Henry, and Kirill Müller. 2020. Dplyr: A Grammar of Data Manipulation. https://CRAN.R-project.org/package=dplyr.
Wickham, Hadley, and Jim Hester. 2020. Readr: Read Rectangular Text Data. https://CRAN.R-project.org/package=readr.
Xie, Yihui. 2014. “Knitr: A Comprehensive Tool for Reproducible Research in R.” In Implementing Reproducible Computational Research, edited by Victoria Stodden, Friedrich Leisch, and Roger D. Peng. Chapman; Hall/CRC. http://www.crcpress.com/product/isbn/9781466561595.
———. 2015. Dynamic Documents with R and Knitr. 2nd ed. Boca Raton, Florida: Chapman; Hall/CRC. https://yihui.org/knitr/.
———. 2021. Knitr: A General-Purpose Package for Dynamic Report Generation in R. https://yihui.org/knitr/.
Xie, Yihui, J. J. Allaire, and Garrett Grolemund. 2018. R Markdown: The Definitive Guide. Boca Raton, Florida: Chapman; Hall/CRC. https://bookdown.org/yihui/rmarkdown.
Xie, Yihui, Christophe Dervieux, and Emily Riederer. 2020. R Markdown Cookbook. Boca Raton, Florida: Chapman; Hall/CRC. https://bookdown.org/yihui/rmarkdown-cookbook.
Zizka, Alexander, Daniele Silvestro, Tobias Andermann, Josue Azevedo, Camila Duarte Ritter, Daniel Edler, Harith Farooq, et al. 2019. “CoordinateCleaner: Standardized Cleaning of Occurrence Records from Biological Collection Databases.” Methods in Ecology and Evolution, no. 10: 744–51. https://doi.org/10.1111/2041-210X.13152.